研究チーム
Team研究チーム
研究項目A01 計画研究:ケモテクノロジーによるユビキチンコードの解読と制御
ケモテクノロジーを利用したタンパク質分解制御
研究代表者 村田 茂穂東京大学大学院薬学系研究科蛋白質代謝学教室 教授 |
|
研究分担者 山野 晃史東京医科歯科大学 難治疾患研究所 機能分子病態学分野 准教授 |
本計画研究の概要
プロテアソームとオートファジーは真核細胞内の主たる分解系であり、生命活動を支えるタンパク質・オルガネラの動的な新陳代謝の中心を担っています。プロテアソームによるタンパク質分解は、高い基質選択性を持ったタンパク質分解であり、様々な生命活動の制御に必須の働きを担っています。オートファジーは、タンパク質凝集体や細胞内オルガネラを隔離膜で取り囲み、リソソームと融合させることによりその内容物を選択的かつ大規模に分解する系です。いずれの分解系においても、その分解するターゲットの選択にユビキチン修飾とその受容体分子が重要な役割を果たしています。これまで、遺伝子改変や発現制御などの手法によりこれら分解系の分子機構と生理機能の解明が進められてきましたが、制御に関与する分子の多くが細胞の生存に必須であり、分解系以外での機能も有しているなどのため、多くの問題が未解明のまま残されていました。本研究ではユビキチンの主たる役割の一つであるタンパク質分解・オルガネラ分解に焦点を当て、細胞内二大分解系であるプロテアソームとオートファジーによる分解過程において、特定のタンパク質間相互作用をタイムリーに喪失させることが可能な化合物や膜透過性ペプチドを利用し、遺伝子改変や発現制御実験では解決できなかった、基質選択性や分解系制御機構の新たな理解の解明を目指します。さらにその成果を活かして、プロテアソーム・オートファジー分解系と分解標的タンパク質・オルガネラを直接連結させるキメラ化合物を作出し、ユビキチン化を介さず直接的かつ選択的にタンパク質を分解誘導する方法論の確立を目指します。
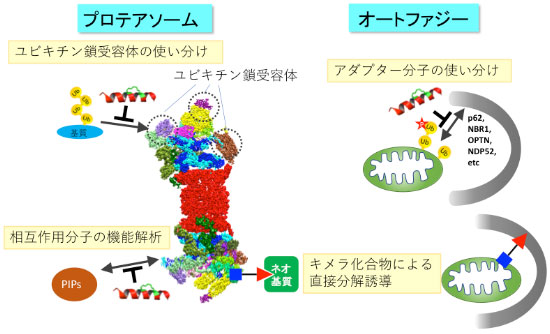
1. ケモテクノロジーによるプロテアソーム制御
プロテアソームユビキチン受容体の基質選別機構の解明、プロテアソームのα-ヘリックスを標的とした相互作用分子機能解析、栄養環境に応答したプロテアソーム分子集合の動的制御の生理的意義の解明を目指します。
2. ケモテクノロジーによるユビキチン選択的オートファジーの制御
オートファジー専用デコーダー分子の基質選別機構の解明、既知のオートファジー経路に依存しないオルガネラ分解機構の探索を行います。
3. キメラ化合物による選択的分解誘導系の開発
プロテアソーム・オートファジーマシナリーと標的タンパク質・オルガネラを連結させるキメラ化合物を作製し、ユビキチン非依存的な選択的分解を誘導する方法論を開発します。
研究概要を示す模式図
本領域での研究成果
- Okatsu K, Sato Y, Yamano K, Matsuda N, Negishi L, Takahashi A, Yamagata A, Goto-Ito S, Mishima M, Ito Y, Oka T, Tanaka K, *Fukai S.
Structural insights into ubiquitin phosphorylation by PINK1.
Sci. Rep. 8, 10382 (2018)
PMID: 29991771 - Wu W, Sahara K, Hirayama S, Zhao X, Watanabe A, Hamazaki J, Yashiroda H, *Murata S.
PAC1‐PAC2 proteasome assembly chaperone retains the core α4–α7 assembly intermediates in the cytoplasm.
Genes to Cells 23, 839–848 (2018)
PMID: 30133132 - Tomaru U, Konno S, Miyajima S, Kimoto R, Onodera M, Kiuchi S, Murata S, Ishizu A, Kasahara M.
Restricted Expression of the Thymoproteasome Is Required for Thymic Selection and Peripheral Homeostasis of CD8+ T Cells.
Cell Rep. 26, 639-651.e2 (2019)
PMID: 30650357 - Ohigashi I, Tanaka Y, Kondo K, Fujimori S, Kondo H, Palin AC, Hoffmann V, Kozai M, Matsushita Y, Uda S, Motosugi R, Hamazaki J, Kubota H, Murata S, Tanaka K, Katagiri T, Kosako H, Takahama Y.
Trans-omics Impact of Thymoproteasome in Cortical Thymic Epithelial Cells.
Cell Rep. 29, 2901-2916.e6 (2019)
PMID: 31775054 - Otsubo R, Mimuro H, Ashida H, Hamazaki J, Murata S, Sasakawa C.
Shigella effector IpaH4.5 targets 19S regulatory particle subunit RPN13 in the 26S proteasome to dampen cytotoxic T lymphocyte activation.
Cell Microbiol. 21, e12974 (2019)
PMID: 30414351 - Bai M, Zhao X, Sahara K, Ohte Y, Hirano Y, Kaneko T, Yashiroda H, Murata S.
In-depth Analysis of the Lid Subunits Assembly Mechanism in Mammals.
Biomolecules 9, 213 (2019)
PMID: 31159305 - Arata Y, Watanabe A, Motosugi R, Iemura S, Natsume T, Mukai K, Taguchi T, Hirayama S, Hamazaki J, Murata S.
FAM48A mediates compensatory autophagy induced by proteasome impairment.
Genes to Cells 24, 559–568 (2019)
PMID: 31210371 - Koyano F, Yamano K, Kosako H, Tanaka K, *Matsuda N.
Parkin recruitment to impaired mitochondria for nonselective ubiquitylation is facilitated by MITOL.
J. Biol. Chem. 294, 10300-10314 (2019)
PMID: 31110043 - Koyano F, Yamano K, Kosako H, Kimura Y, Kimura M, Fujiki Y, Tanaka K, *Matsuda N.
Parkin-mediated ubiquitylation redistributes MITOL/March5 from mitochondria to peroxisomes.
EMBO Rep. 20, e47728 (2019)
PMID: 31602805
プレスリリース - Arata Y, Watanabe A, Motosugi R, Murakami R, Goto T, Hori S, Hirayama S, Hamazaki J, Murata S.
Defective induction of the proteasome associated with T‐cell receptor signaling underlies T‐cell senescence.
Genes to Cells 24, 801–813 (2019)
PMID: 31621149 - Yasuda S, Tsuchiya H, Kaiho A, Guo Q, Ikeuchi K, Endo A, Arai N, Ohtake F, Murata S, Inada T, Baumeister W, Fernández-Busnadiego R, Tanaka K, *Saeki Y.
Stress- and ubiquitylation-dependent phase separation of the proteasome.
Nature 578, 296–300 (2020)
PMID: 32025036
プレスリリース
http://www.igakuken.or.jp/topics/2020/0205.html (都医学研)
https://www.amed.go.jp/news/release_20200206-02.html(AMED)
Highlighted in Cell Res
EurekAlert!
Faculty Opinions - Waku T, Nakamura N, Koji M, Watanabe H, Katoh H, Tatsumi C, Tamura N, Hatanaka A, Hirose S, Katayama H, Tani M, Kubo Y, Hamazaki J, Hamakubo T, Watanabe A, Murata S, Kobayashi A.
NRF3-POMP-20S Proteasome Assembly Axis Promotes Cancer Development via Ubiquitin-Independent Proteolysis of p53 and Retinoblastoma Protein.
Mol. Cell. Biol. 40, e597-19 (2020)
PMID: 32123008 - *Yamano K, Kikuchi R, Kojima W, Hayashida R, Koyano F, Kawawaki J, Shoda T, Demizu Y, Naito M, Tanaka K, *Matsuda N.
Critical role of mitochondrial ubiquitination and the OPTN–ATG9A axis in mitophagy.
J. Cell Biol. 219, e201912144 (2020)
PMID: 32556086 - Hashimoto E, Okuno S, Hirayama S, Arata Y, Goto T, Kosako H, Hamazaki J, *Murata S.
Enhanced O-GlcNAcylation mediates cytoprotection under proteasome impairment by promoting proteasome turnover in cancer cells.
iScience 23, 101299 (2020)
PMID: 32634741 - Kojima W, *Yamano K, Kosako H, Imai K, Kikuchi R, Tanaka K, Matsuda N.
Mammalian BCAS3 and C16orf70 associate with the phagophore assembly site in response to selective and non-selective autophagy.
Autophagy 17, 2011-2036 (2021)
PMID: 33499712
プレスリリース - Takehara Y, Yashiroda H, Matsuo Y, Zhao X, Kamigaki A, Matsuzaki T, Kosako H, Inada T, *Murata S.
The ubiquitination-deubiquitination cycle on the ribosomal protein eS7A is crucial for efficient translation.
iScience 24, 102145 (2021)
PMID: 33665564 - *Kanazawa N, Hemmi H, Kinjo N, Ohnishi H, Hamazaki J, Mishima H, Kinoshita A, Mizushima T, Hamada S, Hamada K, Kawamoto N, Kadowaki S, Honda Y, Izawa K, Nishikomori R, Tsumura M, Yamashita Y, Tamura S, Orimo T, Ozasa T, Kato T, Sasaki I, Fukuda-Ohta Y, Wakaki-Nishiyama N, Inaba Y, Kunimoto K, Okada S, Taketani T, Nakanishi K, Murata S, Yoshiura K, *Kaisho T.
Heterozygous missense variant of the proteasome subunit β-type 9 causes neonatal-onset autoinflammation and immunodeficiency.
Nature Comm. 12, 6819 (2021)
PMID: 34819510 - Watanabe A, Hama K, Watanabe K, Fujiwara Y, Yokoyama K, Murata S, *Takita R.
Controlled tetradeuteration of straight-chain fatty acids: synthesis, application, and insight into the metabolism of oxidized linoleic acid.
Angew Chem Int Ed Engl. e202202779 (2022)
PMID: 35411582 - *Tanahashi N, Komiyama M, Tanaka M, Yokobori Y, Murata S, Tanaka K.
The effect of nutrient deprivation on proteasome activity in 4-week-old mice and 24-week-old mice.
J Nutr Biochem 105, 108993 (2022)
PMID: 35331898 - Hayashida R, Kikuchi R, Imai K, Kojima W, Yamada T, Iijima M, Sesaki H, Tanaka K, *Matsuda N, *Yamano K.
Elucidation of ubiquitin-conjugating enzymes that interact with RBR-type ubiquitin ligases using a liquid-liquid phase separation-based method.
J Biol Chem. 299, 102822 (2023)
PMID: 36563856
関連する代表的な論文
- Yamano K, Fogel AI, Wang C, van der Bliek, AM, Youle RJ.
Mitochondrial Rab GAPs govern autophagosome biogenesis during mitophagy.
eLife 3, e01612 (2014)
PMID: 24569479 - Hamazaki J, Hirayama S, *Murata S.
Redundant roles of Rpn10 and Rpn13 in recognition of ubiquitinated proteins and cellular homeostasis.
PLoS Genet. 11, e1005401 (2015)
PMID: 26222436 - Yamano K, Queliconi BB, Koyano F, Saeki Y, Hirokawa T, Tanaka K, Matsuda N.
Site-specific interaction mapping of phosphorylated ubiquitin to uncover Parkin activation.
J. Biol. Chem. 290, 25199-25211 (2015)
PMID: 26260794 - Sasaki K, Takada K, Ohte Y, Kondo H, Sorimachi H, Tanaka K, *Takahama Y, *Murata S.
Thymoproteasomes produce unique peptide motifs for positive selection of CD8+ T cells.
Nat. Commun. 6, 7484 (2015)
PMID: 26099460 - Yamano K, Matsuda N, Tanaka K.
The Ubiquitin signal and autophagy: an orchestrated dance leading to mitochondrial degradation.
EMBO Rep. 17, 300-316 (2016)
PMID: 26882551 - Koizumi S, Irie T, Hirayama S, Sakurai Y, Yashiroda H, Naguro I, Ichijo H, Hamazaki J, *Murata S.
The aspartyl protease DDI2 activates Nrf1 to compensate for proteasome dysfunction.
elife 5, e18357 (2016)
PMID: 27528193 - Lu X, Nowicka U, Sridharan V, Liu F, Randles L, Hymel D, Dyba M, Tarasov SG, Tarasova NI, Zhao XZ, Hamazaki J, Murata S, Burke TR Jr, *Walters KJ.
Structure of the Rpn13-Rpn2 complex provides insights for Rpn13 and Uch37 as anticancer targets.
Nat. Commun. 8, 15540 (2017)
PMID: 28598414 - Yamano K, Wang C, Sarraf SA, Münch C, Kikuchi R, Noda NN, Hizukuri Y, Kanemaki MT, Harper W, Tanaka K, Matsuda N, Youle RJ.
Endosomal Rab cycles regulate Parkin-mediated mitophagy.
eLife 7, e31326 (2018)
PMID: 29360040 - Uechi H, Kuranaga E, Iriki T, Takano K, Hirayama S, Miura M, Hamazaki J, *Murata S.
Ubiquitin-Binding Protein CG5445 Suppresses Aggregation and Cytotoxicity of Amyotrophic Lateral Sclerosis-linked TDP-43 in Drosophila.
Mol. Cell. Biol. 38, e00195-17 (2018)
PMID: 29109084 - *Hirayama S, Sugihara M, Morito D, Iemura S, Natsume T, Murata S, *Nagata K.
Nuclear export of ubiquitinated proteins via the UBIN-POST system.
Proc. Natl. Acad. Sci. U.S.A. 115, E4199-E4208 (2018)
PMID: 29666234 - Murata S, Takahama Y, Kasahara M, Tanaka K.
The immunoproteasome and thymoproteasome: functions, evolution and human disease.
Nat. Immunol. 19,923-931 (2018)
PMID: 30104634





