森戸 大介
ケモテクノロジーで探るDNAメチル化部位特異的なユビキチンシグナル機能とその制御
 |
研究代表者 西山 敦哉東京大学 医科学研究所 癌防御シグナル分野 准教授 |
|---|
研究概要
DNAメチル化は様々な生命現象に重要な役割を果たすエピジェネティクス修飾であり、その脱制御はがんをはじめとする様々な疾患を引き起こすことが知られています。ゲノム上のDNAメチル化模様はDNA複製時に正確に娘染色体に継承される必要があり、これは片鎖メチル化DNA特異的結合タンパク質UHRF1と片鎖メチル化DNAの両鎖メチル化DNAへの変換を司るDNAメチル化酵素DNMT1を中核とするDNAメチル化維持機構によって制御されていますが、その分子機構はまだ完全には理解されていません。
私たちはDNAメチル化継承において、UHRF1がE3ユビキチンリガーゼとして働くことで生成されるヒストンH3のマルチプルモノユビキチン化がDNAメチル化酵素DNMT1によって認識されることが重要であることを明らかにしてきました。さらに、新たなDNMT1結合タンパク質としてPCNA結合タンパク質の一つであるPAF15を同定し、PAF15がヒストンH3同様なマルチプルモノユビキチン化を受けること、そしてマルチプルモノユビキチン化PAF15がS期前期における維持DNAメチル化に重要な役割を果たしていることを見出しています。
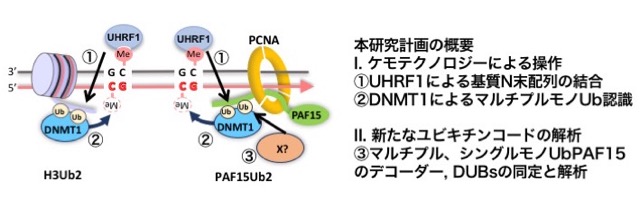
本研究計画では維持DNAメチル化の過程で重要な2つの分子間結合、即ち(1)E3ユビキチンリガーゼUHRF1によるPHDドメインを介した基質タンパク質N末配列との相互作用、(2)DNMT1によるマルチプルモノユビキチンの認識と結合、をケモテクノロジーにより操作することをめざします。これまでの解析によって得られた知見をベースとして、複合体形成を阻害する活性を持つステープルペプチドや小分子化合物の取得に取り組みます。また並行して、PAF15タンパク質のユビキチン化制御を制御する脱ユビキチン化酵素やデコーダータンパク質についても解析を進める予定です。
研究概要を示す模式図
本領域での研究成果
- Mishima Y, Brueckner L, Takahashi S, Kawakami T, Otani J, Shinohara A, Takeshita K, Garvilles RG, Watanabe M, Sakai N, Takeshima H, Nachtegael C, Nishiyama A, Nakanishi M, Arita K, Nakashima K, Hojo H, Suetake I.
Enhanced processivity of Dnmt1 by monoubiquitinated histone H3.
Genes to Cells 25, 22-32 (2020)
PMID: 31680384 - *Nishiyama A, Mulholland C, Bultmann S, Kori A, Endo A, Saeki Y, Qin W, Trummer C, Chiba Y, Yokoyama H, Kumamoto S, Kawakami T, Hojo H, Nagae G, Aburatani H, Tanaka K, *Arita K, *Leonhardt H, *Nakanishi M.
Two distinct modes of DNMT1 recruitment ensure the stable maintenance DNA methylation.
Nat. Commun. 11, 1222 (2020)
PMID: 32144273
プレスリリース
Faculty Opinions - Mulholland CB, Nishiyama A, Ryan J, Nakamura R, Yiğit M, Glück IM, Trummer C, Qin W, Bartoschek MD, Traube FR, Parsa E, Ugur E, Modic M, Acharya A, Stolz P, Ziegenhain C, Wierer M, Enard W, Carell T, Lamb DC, Takeda H, Nakanishi M, Bultmann S, *Leonhardt H.
Recent evolution of a TET-controlled and DPPA3/STELLA-driven pathway of passive DNA demethylation in mammals.
Nat. Commun. 11, 5972 (2020)
PMID: 33235224
代表的な論文
- *Nishiyama A, Yamaguchi L, Sharif J, Johmura Y, Kawamura T, Nakanishi K, Shimamura S, Arita K, Kodama T, Ishikawa F, Koseki H, *Nakanishi M.
Uhrf1-dependent H3K23 ubiquitylation couples maintenance DNA methylation and replication.
Nature 502,249-253 (2013)
PMID: 24013172 - Nishiyama A, Yamaguchi L, *Nakanishi M.
Regulation of maintenance DNA methylation via histone ubiquitylation.
J. Biochem. 159,9-15 (2016)
PMID: 26590302 - Misaki T, Yamaguchi L, Sun J, Orii M, *Nishiyama A, *Nakanishi M.
The replication foci targeting sequence (RFTS) of DNMT1 functions as a potent histone H3 binding domain regulated by autoinhibition.
Biochem. Biophys. Res. Commun. 470,741-747 (2016)
PMID: 26774338 - Yamaguchi L, *Nishiyama A, Misaki T, Johmura Y, Ueda J, Arita K, Nagao K, Obuse C, *Nakanishi M.
Usp7-dependent histone H3 deubiquitylation regulates maintenance of DNA methylation.
Sci. Rep. 7,55 (2017)
PMID: 28246399 - Ishiyama S, Nishiyama A, Saeki Y, Moritsugu K, Morimoto D, Yamaguchi L, Arai N, Matsumura R, Kawakami T, Mishima Y, Hojo H, Shimamura S, Ishikawa F, Tajima S,Tanaka K, Ariyoshi M, Shirakawa M, Ikeguchi M, Kidera A, *Suetake I, *Arita K, *Nakanishi M.
Structure of the Dnmt1 Reader Module Complexed with a Unique Two-Mono-Ubiquitin Mark on Histone H3 Reveals the Basis for DNA Methylation Maintenance.
Mol. Cell 68,350-360(2017)
PMID: 29053958





