西山 敦哉
5-aza-dCTPによる新たなDNA維持メチル化制御機構の解明
 |
研究代表者 西山 敦哉東京大学 医科学研究所 癌防御シグナル分野 准教授 |
|---|
研究概要
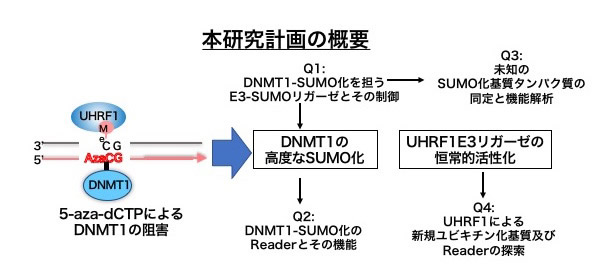
DNAメチル化は、様々な生命現象に重要な役割を果たすエピジェネティクス修飾であり、その脱制御は様々な疾患の原因となります。ゲノム上のDNAメチル化パターンは、DNA複製時に維持型DNAメチル化酵素DNMT1によって、娘DNAに継承されます。近年、DNMT1のDNAメチル化部位への局在と活性化において、E3ユビキチンリガーゼUHRF1によって触媒されるマルチプルモノユビキチン化が重要な役割を果たしていることが明らかとなってきました。5-aza-dCTPはDNA複製時にゲノムDNAに取り込まれると、DNAメチル酵素と不可逆的な架橋結合を形成することでDNA脱メチル化を誘導する核酸アナログであり、抗がん剤としても認可されている低分子化合物です。しかし、5-aza-dCTPとDNMTによって形成されるDNAータンパク質架橋が細胞内で、どのようなプロセスにより処理されていて、UHRF1によるユビキチンシグナルにどのような影響を与えるのかは明らかになっていません。本研究はDNA複製に伴うDNAメチル化維持の過程を試験管内で再現可能なツメガエル卵抽出液を実験系に用いて、この問題に取り組みます。まず、私達は、試験管内系において、5-aza-dCTPがDNMT1やUHRF1の過剰なクロマチンへの集積を引き起こすとともに、UHRF1の下流で働くユビキチンシグナル経路を強く活性化すること、さらにDNMT1の高度なSUMO化を誘導することを見出しました。そこで、このような条件下で特異的にクロマチンに集積するタンパク質を同定するために、5-aza-dCTP処理に伴い、SUMO依存的に、あるいは非依存的にクロマチンに集積するタンパク質についてChromatin Mass spectrometry (CHROMASS) 法を用いて解析し、約20種のタンパク質の同定に成功しました。これらのタンパク質はUHRF1の下流で働く未知のDNAメチル化制御因子、あるいはDNMT1-DNA架橋の制御因子として働く可能性があり、今後その作用機序と分子機能を明らかにしていく予定です。
研究概要を示す模式図
本領域での研究成果
- Kumamoto S, *Nishiyama A, Chiba Y, Miyashita R, Konishi C, Azuma Y, *Nakanishi M.
HPF1-dependent PARP activation promotes LIG3-XRCC1-mediated backup pathway of Okazaki fragment ligation.
- Kori S, Shibahashi Y, Ekimoto T, Nishiyama A, Yoshimi S, Yamaguchi K, Nagatoishi S, Ohta M, Tsumoto K, Nakanishi M, Defossez PA, Ikeguchi M, *Arita K.
Structure-based screening combined with computational and biochemical analyses identified the inhibitor targeting the binding of DNA Ligase 1 to UHRF1.
- Kikuchi A, Onoda H, Yamaguchi K, Kori S, Matsuzawa S, Chiba Y, Tanimoto S, Yoshimi S, Sato H, Yamagata A, Shirouzu M, Adachi N, Sharif J, Koseki H, Nishiyama A, Nakanishi M, Defossez PA , Arita K.
Structural basis for activation of DNMT1.
Nature Communications 13, 7130 (2022)
PMID: 36414620 - Hata K, Kobayashi N, Sugimura K, Qin W, Haxholli D, Chiba Y, Yoshimi S, Hayashi G, Onoda H, Ikegami T, Mulholland CB, Nishiyama A, Nakanishi M, Leonhardt H, Konuma T, Arita K.
Structural basis for the unique multifaceted interaction of DPPA3 with the UHRF1 PHD finger.
Nucleic Acids Res. 50, 12527-12542 (2022)
PMID: 36420895 - Miyashita R, *Nishiyama A, Qin W, Chiba Y, Kori S, Kato N, Konishi C, Kumamoto S, Kozuka-Hata H, Oyama M, Kawasoe Y, Tsurimoto T, Takahashi TS, Leonhardt H, Arita K, *Nakanishi M.
The termination of UHRF1-dependent PAF15 ubiquitin signaling is regulated by USP7 and ATAD5.
eLife. AOP (2023)
PMID: 36734974
代表的な論文
- *Nishiyama A, Yamaguchi L, Sharif J, Johmura Y, Kawamura T, Nakanishi K, Shimamura S, Arita K, Kodama T, Ishikawa F, Koseki H, *Nakanishi M.
Uhrf1-dependent H3K23 ubiquitylation couples maintenance DNA methylation and replication.
Nature 502,249-253 (2013)
PMID: 24013172 - Nishiyama A, Yamaguchi L, *Nakanishi M.
Regulation of maintenance DNA methylation via histone ubiquitylation.
J. Biochem. 159,9-15 (2016)
PMID: 26590302 - Yamaguchi L, *Nishiyama A, Misaki T, Johmura Y, Ueda J, Arita K, Nagao K, Obuse C, *Nakanishi M.
Usp7-dependent histone H3 deubiquitylation regulates maintenance of DNA methylation.
Sci. Rep. 7,55 (2017)
PMID: 28246399 - Ishiyama S, Nishiyama A, Saeki Y, Moritsugu K, Morimoto D, Yamaguchi L, Arai N, Matsumura R, Kawakami T, Mishima Y, Hojo H, Shimamura S, Ishikawa F, Tajima S,Tanaka K, Ariyoshi M, Shirakawa M, Ikeguchi M, Kidera A, *Suetake I, *Arita K, *Nakanishi M.
Structure of the Dnmt1 Reader Module Complexed with a Unique Two-Mono-Ubiquitin Mark on Histone H3 Reveals the Basis for DNA Methylation Maintenance.
Mol. Cell 68,350-360 (2017)
PMID: 29053958 - *Nishiyama A, Mulholland C, Bultmann S, Kori A, Endo A, Saeki Y, Qin W, Trummer C, Chiba Y, Yokoyama H, Kumamoto S, Kawakami T, Hojo H, Nagae G, Aburatani H, Tanaka K, *Arita K, *Leonhardt H, *Nakanishi M.
Two distinct modes of DNMT1 recruitment ensure the stable maintenance DNA methylation.
Nat. Commun. 11, 1222 (2020)
PMID: 32144273





