松尾 芳隆
ケモテクノロジーを活用したリボソームユビキチンコードの解読と制御
 |
研究代表者 松尾 芳隆東京大学 医科学研究所 RNA制御学分野 准教授 |
|---|
研究概要
タンパク質の合成を司るリボソームの機能は、mRNAに示された遺伝情報をタンパク質へ変換するという機械的なものとして認識されていましたが、近年、リボソーム自身のユビキチン化修飾によって制御される様々な現象が明らかになってきています。
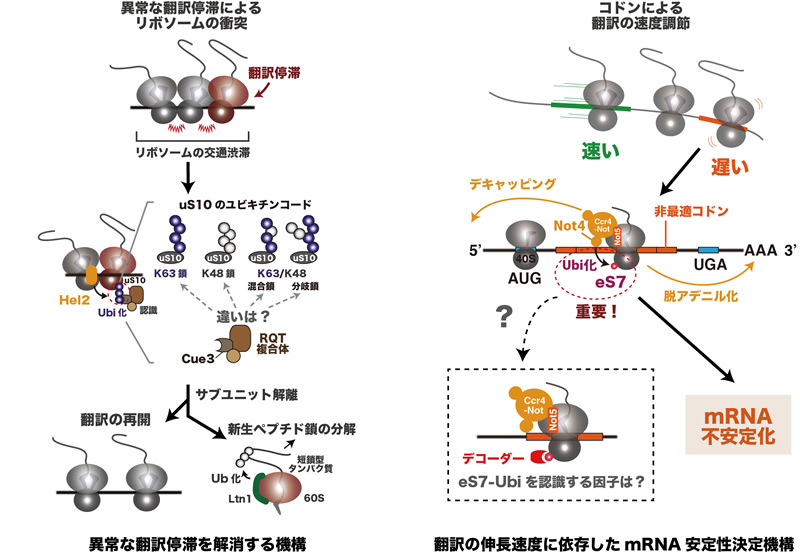
わたしたちは、異常な翻訳停滞を認識し、リボソームタンパク質uS10をユビキチン化するセンサータンパク質“Hel2”(E3)や、新生ペプチド鎖を分解する品質管理機構(Ribosome-associated Quality Control: RQC)の誘導起点となるサブユニット解離に必須な“RQT複合体”(デコーダー)を発見しました。さらに、翻訳停滞からuS10のユビキチン化を介したサブユニット解離までの一連の反応を試験管内で再現することで、RQCが誘導される分子機構の詳細を明らかにしました(図:左)。
また、わたしたちは、コドンの最適化によって調整される“翻訳の伸長速度”を監視することで、mRNAの安定性を決定する因子として、“Ccr4-Not複合体”を見出しました。Ccr4-Not複合体は、構成因子であるNot5を介して非最適コドンを翻訳するリボソームに結合し、mRNAの不安定化を誘導します。また、この機構には、Ccr4-Not複合体の構成因子である“Not4”(E3)によるリボソームタンパク質eS7へのユビキチン化が必須であることも明らかになりました(図:右)。
このように、細胞内では、翻訳途中のリボソームの動体を監視し、その運命を決定する機構が複数存在しますが、いずれについてもリボソーム自身のユビキチン化が起点になっています。一方で、ユビキチン鎖の認識・作動機構の理解は不十分なままです。
本研究では、上述した2つの機構:【1】異常な翻訳停滞を解消する品質管理機構と【2】翻訳の伸長速度に依存したmRNAの安定性決定機構において重要な役割を果たすリボソームのユビキチン化に注目し、部位特異的、かつユビキチン鎖特異的な作動機構の解明を進めます。また、並行して、各ユビキチン鎖の認識を特異的に調節するケミカルツールを開発し、リボソームユビキチンコードの制御を目指します。
研究概要を示す模式図
本領域での研究成果
- Li S, Ikeuchi K, Kato M, Buschauer R, Sugiyama T, Adachi S, Kusano H, Natsume T, Berninghausen O, Matsuo Y, Becker T, *Beckmann R, *Inada T.
Sensing of individual stalled 80S ribosomes by Fap1 for non-functional rRNA turnover.
Mol Cell. 82, 3424-3437 (2022)
PMID: 36113412
プレスリリース - Narita M, Denk T, Matsuo Y, Sugiyama T, Kikuguchi C, Ito S, Sato N, Suzuki T, Hashimoto S, Machova I, Tesina P, *Beckmann R, *Inada T.
A distinct human disome collision interface harbors K63-linked polyubiquitination of uS10 to trigger hRQT-mediated subunit dissociation.
Nat Commun. 13, 6411 (2022)
PMID: 36302773
プレスリリース - *Matsuo Y, Uchihashi T, *Inada T.
Decoding of the ubiquitin code for clearance of colliding ribosomes by the RQT complex.
Nat Commun. 14, 79 (2023)
PMID: 36627279
プレスリリース - Tomomatsu S, Watanabe A, Tesina P, Hashimoto S, Ikeuchi K, Li S, Matsuo Y, Beckmann R, *Inada T.
Two modes of Cue2-mediated mRNA cleavage with distinct substrate recognition initiate No-go decay.
Nucleic Acids Res. 51, 253-270 (2023)
PMID: 36583309
プレスリリース - *Tesina P, Ebine S, Buschauer R, Thoms M, Matsuo Y, *Inada T, *Beckmann R.
Molecular basis of eIF5A-dependent CAT tailing in eukaryotic ribosome-associated quality control.
Mol Cell. 83, 607-621 (2023)
PMID: 36804914 - Best K, Ikeuchi K, Kater L, Best D, Musial J, Matsuo Y, Berninghausen O, Becker T, *Inada T, *Beckmann R.
Structural basis for clearing of ribosome collisions by the RQT complex.
Nat Commun. 14, 921 (2023)
PMID: 36801861
代表的な論文
- Matsuo Y, Granneman S, Thoms M, Manikas RG, Tollervey D, *Hurt E.
- Matsuo Y, Ikeuchi K, Saeki Y, Iwasaki S, Schmidt C, Udagawa T, Sato F, Tsuchiya H, Becker T, Tanaka K, Ingolia NT, Beckmann R, *Inada T.
- Matsuo Y, Tesina P, Nakajima S, Mizuno M, Endo A, Buschauer R, Cheng J, Shounai O, Ikeuchi K, Saeki Y, Becker T, *Beckmann R, *Inada T.
- Buschauer R, Matsuo Y, Sugiyama T, Chen YH, Alhusaini N, Sweet T, Ikeuchi K, Cheng J, Matsuki Y, Nobuta R, Gilmozzi A, Berninghausen O, Tesina P, Becker T, *Coller J, *Inada T, *Beckmann R.
- *Matsuo Y ,*Inada T.
Coupled GTPase and remodelling ATPase activities form a checkpoint for ribosome export.
Nature. 505, 112-116 (2014)
PMID: 24240281
Ubiquitination of stalled ribosome triggers ribosome-associated quality control.
Nat. Commun. 8, 159 (2017)
PMID: 28757607
RQT complex dissociates ribosomes collided on endogenous RQC substrate SDD1.
Nat. Struct. Mol. Biol. 27, 323-332 (2020)
PMID: 32203490
The Ccr4-Not complex monitors the translating ribosome for codon optimality.
Science. 368, eaay6912 (2020)
PMID: 32299921
The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway.
Cell Rep. 34, 108877 (2021)
PMID: 33761353





