畠山 鎮次
TRIM型ユビキチンリガーゼの物性と動作原理の解析
 |
研究代表者 畠山 鎮次北海道大学大学院医学研究院医化学教室 教授 |
|---|
研究概要
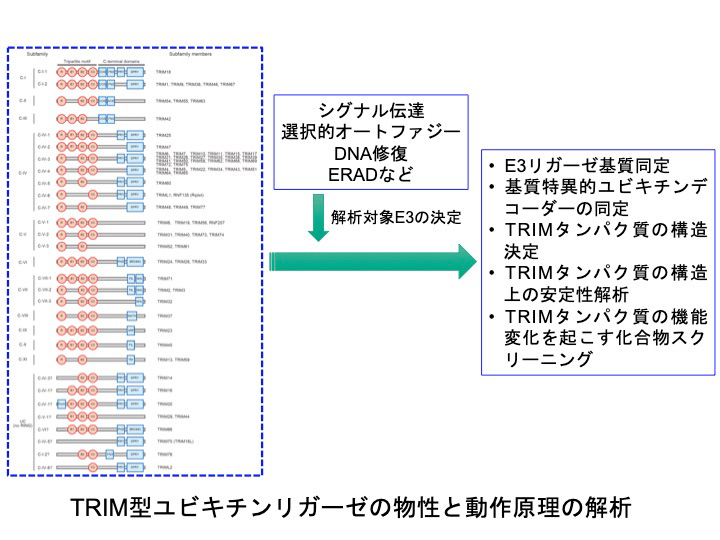
TRIMファミリーはRING型E3ユビキチンリガーゼの一つのファミリー(ヒトでは約70種)であり、様々な機能(がん、免疫、発生、オートファジーなどの制御)に関与することが報告されています。本研究の目的は、TRIMタンパク質の「タンパク質」としての物理化学的な性質(物性)を解析することで機能との関連性を検討し、将来的には創薬研究への知見を集積することであります。
TRIMタンパク質の機能を解明するためにはまず、その基質タンパク質を同定することと、TRIMタンパク質の構造を決定することが必要です。それらの情報および疾患との関係をもとに、対象とするTRIMタンパク質を決定し、機能変化をもたらす化合物スクリーニングを遂行することは、臨床上、重要となります。そのためには、特異性の高いTRIMタンパク質の基質タンパク質及びユビキチンデコーダーの同定法(TUBE-E3法)の樹立を進めます。また同時に、TRIMタンパク質の構造生物学的解析を進めることが必要となります。本計画で対象とするE3リガーゼ候補としては、「シグナル伝達」「選択的オートファジー」「DNA修復」「ERAD」等に関与すると考えられているTRIMタンパク質を中心に解析を進めます。
プロテオミクスや構造生物学、ケミカルバイオロジーの観点から、以下の方法により、上記の対象E3の解析を進めることで「化学ツール開発」及び「創薬」に役立つ基盤的知見を集積します。
- E3リガーゼ基質同定:基質タンパク質を効果的に同定するプロテオミクス解析(TUBE-E3法)を実用レベルまで完成させ、上記の対象E3リガーゼ(TRIMタンパク質)に関して、基質同定を進めます。
- 基質特異的ユビキチンデコーダーの同定:TUBE-E3法を応用することで、基質特異的ユビキチンデコーダーの同定を進めます。
- TRIMタンパク質の構造決定:まだ構造決定が報告されていない上記のTRIMタンパク質の構造決定を進めます。リコンビナントタンパク質を作製し、X線結晶構造解析、NMR、クライオ電子顕微鏡法(cryo-EM)などを使用します。
- TRIMタンパク質の構造上の安定性解析:構造生物学的解析にも利用されるリコンビナントタンパク質を使い、サーマルシフトアッセイを遂行することで、タンパク質としての安定性のデータを得ます。
- TRIMタンパク質の機能変化を起こす化合物スクリーニング:ルシフェラーゼアッセイ等で機能調節化合物が同定できた場合は、ユビキチン関連創薬研究に利用します。
研究概要を示す模式図
本領域での研究成果
- Takahashi H, Ranjan A, Chen S, Suzuki H, Shibata M, Hirose T, Hirose H, Sasaki K, Abe R, Chen K, He Y, Zhang Y, Takigawa I, Tsukiyama T, Watanabe M, Fujii S, Iida M, Yamamoto J, Yamaguchi Y, Suzuki Y, Matsumoto M, Nakayama KI, Washburn M, Saraf A, Florens L, Sato S, Tomomori-Sato C, Conaway RC, Conaway JW, *Hatakeyama S.
The role of Mediator and Little Elongation Complex in transcription termination.
Nature Commun. 11, 1063 (2020)
PMID: 32102997 - Sugawara E, *Kato M, Kudo Y, Lee W, Hisada R, Fujieda Y, Oku K, Bohgaki T, Amengual O, Yasuda S, Onodera T, Hatakeyama S, Atsumi T.
Autophagy promotes citrullination of VIM (vimentin) and its interaction with major histocompatibility complex class II in synovial fibroblasts.
Autophagy 16, 946-955(2020)
PMID: 31486697 - Sugisawa E, Takayama Y, Takemura N, Kondo T, Hatakeyama S, Kumagai Y, Sunagawa M, Tominaga M, *Maruyama K.
RNA sensing by gut Piezo1 is essential for systematic serotonin synthesis.
Cell 182, 609-624 (2020)
PMID: 32640190
代表的な論文
- Masuda Y, Takahashi H, Sato S, Tomomori-Sato C, Saraf A, Washburn WP, Florens L, Conaway RC, Conaway JW,*Hatakeyama S.
TRIM29 regulates the assembly of DNA repair proteins into damaged chromatin.
Nat. Commun. 6, 7299 (2015)
PMID: 26095369 - Mizushima W, Takahashi H, Watanabe M, Kinugawa S, Matsushima S, Takada S, Yokota T, Furihata T, Matsumoto J, Tsuda M, Chiba I, Nagashima S, Yanagi S, Matsumoto M, Nakayama KI, Tsutsui H, *Hatakeyama S.
The novel heart-specific RING finger protein 207 is involved in energy metabolism in cardiomyocytes.
J. Mol. Cell. Cardiol. 100, 43-53 (2016)
PMID: 27677939 - *Hatakeyama S.
TRIM Family Proteins: Roles in Autophagy, Immunity and Carcinogenesis.
Trends Biochem. Sci. 42, 297-311 (2017)
PMID: 28118948 - Sang Y, Li Y, Song L, Alvarez AA, Zhang W, Lv D, Tang J, Liu F, Chang Z, Hatakeyama S, Hu B, Cheng S,*Feng H.
TRIM59 promotes gliomagenesis by inhibiting TC45 dephosphorylation of STAT3.
Cancer Res. 78, 1792-1804 (2018)
PMID: 29386185 - *Yanagi T, Watanabe M, Hata M, Kitamura S, Imafuku K, Yanagi H, Homma MD A, Wang L, Takahashi H, Shimizu H, *Hatakeyama S.
Loss of TRIM29 alters keratin distribution to promote cell invasion in squamous cell carcinoma.
Cancer Res. 78, 6795-6806 (2018)
PMID: 30389700
プレスリリース





