有田 恭平
UHRF1によるマルチモノユビキチン化反応の構造基盤
 |
研究代表者 有田 恭平横浜市立大学大学院 生命医科学研究科 構造生物学研究室 准教授 |
|---|
研究概要
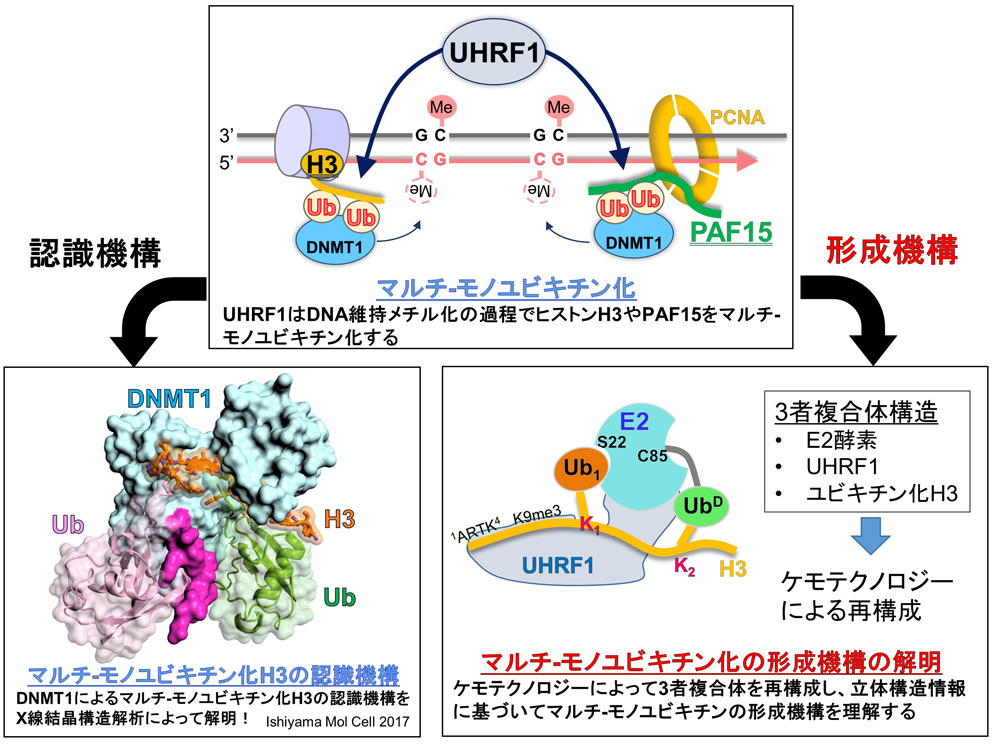
ゲノムDNAのCG配列中のシトシンに起こるDNAメチル化は遺伝子の不活化に関与します。分化した細胞では、細胞の種類に応じて発現する遺伝子が異なりますが、これは各細胞に刻まれるDNAメチル化パターンの違いに起因します。従って、分化した細胞の形質を維持するために、我々の細胞のDNAメチル化は塩基配列と同様に複製され継承されていきます。これをDNA維持メチル化といいます。このDNA維持メチル化にはユビキチン化E3リガーゼ活性をもつUHRF1とDNAメチル化酵素DNMT1の2つのタンパク質が必須の働きをします。DNA維持メチル化は2つの異なる機構が働いていると考えられています。
1つ目はヒストンH3のユビキチン化を介した経路です。UHRF1はヌクレオソームを形成するヒストンH3のK14, K18, K23をマルチモノユビキチン化します。このマルチモノユビキチン化H3をDNMT1が認識することによって、DNMT1はヘミメチル化DNAに呼び込まれ、新生鎖をメチル化します。もう一つは、複製因子PAF15のユビキチン化を介した経路です。UHRF1は複製サイトに局在するPAF15をマルチモノユビキチン化し、DNMT1がヘミメチル化DNAに呼び込まれます。
興味深いことにDNA維持メチル化の過程でDNMT1を働く場所に呼び込むシグナルとなるヒストンH3やPAF15のユビキチン化は、これまでによく研究されてきたポリユビキチン化ではなく、複数のリジン残基がモノユビキチン化されるマルチモノユビキチン化でした。私たちはこれまでにDNMT1がどのようにマルチモノユビキチン化H3を認識するかを明らかにしました。しかし、UHRF1がどのような作用機序でH3やPAF15を段階的にマルチモノユビキチン化するかは全くわかっていません。そこで本研究では、UHRF1によるH3やPAF15のマルチモノユビキチン反応機構の構造基盤を解明するために、ケモテクノロジーを駆使してE2酵素:UHRF1 (E3):基質 (H3またはPAF15)の3者複合体を再構成します。その立体構造をX線結晶構造解析やクライオ電子顕微鏡解析によって明らかにし、立体構造情報に基づいてマルチモノユビキチン化の形成機構を明らかにします。
研究概要を示す模式図
代表的な論文
- Arita K, Ariyoshi M, Tochio H, Nakamura Y, *Shirakawa M.
Recognition of hemi-methylated DNA by the SRA protein UHRF1 by a base-flipping mechanism.
Nature 455, 818-821 (2008)
PMID: 18772891 - Arita K, Isogai S, Oda T. Unoki M, Sugita K, Sekiyama N, Kuwata K, Hamamoto R, Tochio H, Sato M, Ariyoshi M, *Shirakawa M.
Recognition of modification status on a histone H3 tail by linked histone reader modules of the epigenetic regulator UHRF1.
Proc. Natl. Acad. Sci. USA. 109, 12950-12955 (2012)
PMID: 22837395 - Ferry L, Fournier A, Tsusaka T, Adelmant G, Shimazu T, Matano S, Kirsh O, Amouroux R, Dohmae N, Suzuki T, Filion GJ, Deng W, de Dieuleveult M, Fritsch L, Kudithipudi S, Jeltsch A, Leonhardt H, Hajkova P, Marto JA, Arita K, *Shinkai Y, *Defossez PA.
Methylation of DNA Ligase 1 by G9a/GLP recruits UHRF1 to replicating DNA and regulates DNA methylation.
Mol. Cell 67, 550-565 (2017)
PMID: 28803780 - Ishiyama S, Nishiyama A, Saeki Y, Moritsugu K, Morimoto D, Yamaguchi L, Arai N, Matsumura R, Kawakami T, Mishima Y, Hojo H, Shimamura S, Ishikawa F, Tajima S, Tanaka K, Ariyoshi M, Shirakawa M, Ikeguchi M, Kidera A, Suetake I, *Arita K, *Nakanishi M.
Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance.
Mol. Cell 68, 350-360 (2017)
PMID: 29053958 - Kori S, Ferry L, Matano S, Jimenji T, Kodera N, Tsusaka T, Matsumura R, Oda T, Sato M, Dohmae N, Ando T, Shinkai Y, *Defossez PA, *Arita K.
Structure of the UHRF1 Tandem Tudor Domain Bound to a Methylated Non-histone Protein, LIG1, Reveals Rules for Binding and Regulation.
Structure 27, 485-496 (2019)
PMID: 30639225





