選択的ユビキチン識別機構の構造生物学


研究概要:
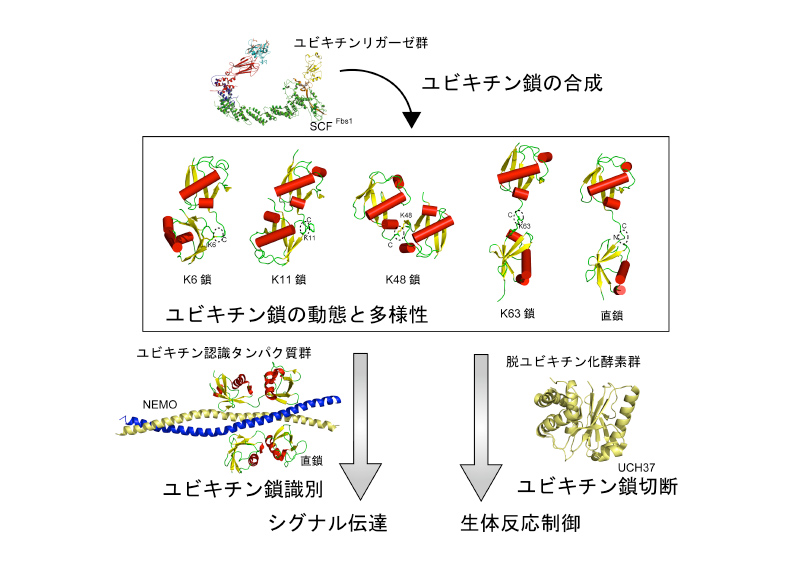
多彩な生命現象の制御において中核的な役割を果たすユビキチン化修飾は分子内に7 個存在するリジン(K6、K11、K27、K29、K33、K48、K63)、およびN末端のメチオニン(M1)を介した8 種のユビキチン間の連結により、ポリユビキチン鎖としての構造的多様性を獲得し、タンパク質機能を調節しています。このユビキチン化修飾による膨大な情報(ユビキチンコード)はユビキチン鎖を認識する識別タンパク質、合成を行うユビキチンリガーゼ(E3)、除去の役割を持った脱ユビキチン化酵素に加え、多様な連結様式によりタンパク質機能を調節するユビキチン鎖により制御されています。この多様なユビキチン鎖を介したタンパク質の相互作用の特異性と機能制御のメカニズムの詳細を理解するためには、機能状態のタンパク質およびタンパク質-タンパク質複合体の三次元構造情報が必要不可欠です。本研究ではユビキチン修飾経路の各段階において選択的なユビキチン識別に関わるタンパク質の構造解析を行い、それらによる調節機構を、立体構造を基盤とした構造生物学的側面から解析します。
関連する代表的な論文:
(水島)
- Nishide, A., Kim, M., Takagi, K., Himeno, A., Sanada, T., Sasakawa, C., and Mizushima, T. (2013) Structural basis for the recognition of Ubc13 by the Shigella flexneri effector OspI. J. Mol. Biol. 425, 2623-2631.
- Sanada, T., Kim, M., Mimuro, H., Suzuki, M., Ogawa, M., Oyama, A., Ashida, H., Kobayashi, T., Koyama, T., Nagai, S., Shibata, Y., Gohda, J., Inoue, J., Mizushima, T., and Sasakawa, C. (2012) The Shigella flexneri effector OspI deamidates UBC13 to dampen the inflammatory response. Nature 483, 623-626.
- Hirano, T., Serve, O., Yagi-Utsumi, M., Takemoto, E., Hiromoto, T., Satoh, T., Mizushima, T., and Kato, K. (2011) Conformational dynamics of wild-type Lys-48-linked diubiquitin in solution. J. Biol. Chem. 286, 37496-37502.
- Mizushima, T., Yoshida, Y., Kumanomidou, T., Hasegawa, Y., Suzuki, A., Yamane, T., and Tanaka, K. (2007) Structural basis for the selection of glycosylated substrates by SCFFbs1 ubiquitin ligase. Proc. Natl. Acad. Sci. USA 104, 5777-5781.
- Mizushima, T., Hirao, T., Yoshida, Y., Lee, S.J., Chiba, T., Iwai, K., Yamaguchi, Y., Kato, K., Tsukihara, T., and Tanaka, K. (2004) Structural basis of sugar-recognizing ubiquitin ligase. Nat. Struct. Mol. Biol. 11, 365-370.
(加藤)
- Kuwabara, N., Minami, R., Yokota, N., Matsumoto, H., Senda, T., Kawahara, H., and Kato, R. (2015) Structure of a BAG6 (Bcl-2-associated Athanogene 6)-Ubl4a (Ubiquitin-like Protein 4a) Complex Reveals a Novel Binding Interface That Functions in Tail-anchored Protein Biogenesis. J. Biol. Chem. 290, 9387-9398.
- Fujita, H., Rahighi, S., Akita, M., Kato, R., Sasaki, Y., Wakatsuki, S., and Iwai, K. (2014) Mechanism underlying IKK activation mediated by the linear ubiquitin chain assembly complex (LUBAC). Mol. Cell. Biol. 34, 1322-1335.
- Rohaim, A., Kawasaki, M., Kato, R., Dikic, I., and Wakatsuki, S. (2012) Structure of a compact conformation of linear diubiquitin. Acta Crystallogr. D 68, 102-108.
- Rahighi, S., Ikeda, F., Kawasaki, M., Akutsu, M., Suzuki, N., Kato, R., Kensche, T., Uejima, T., Bloor, S., Komander, D., Randow, F., Wakatsuki, S., and Dikic, I. (2009) Specific recognition of linear ubiquitin chains by NEMO is important for NF-kappaB activation. Cell 136, 1098-1109.
- Hirano, S., Kawasaki, M., Ura, H., Kato, R., Raiborg, C., Stenmark, H., and Wakatsuki, S. (2006) Double-sided ubiquitin binding of Hrs-UIM in endosomal protein sorting. Nat. Struct. Mol. Biol. 13, 272-277.