選択的なポリユビキチン鎖検出・定量法の開発

研究概要:
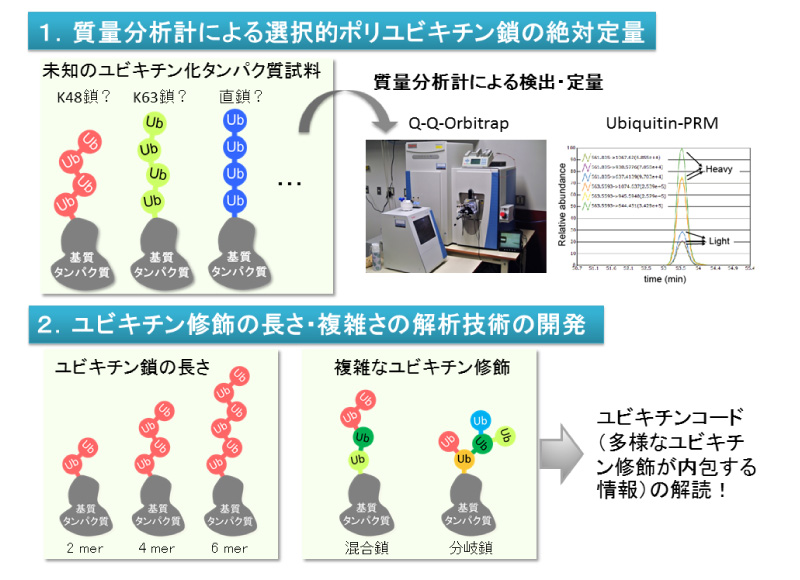
ユビキチンによるタンパク質の翻訳後修飾は、分解のみならずシグナル伝達やタンパク質の輸送など広汎な生命機能を制御することが明確となってきました。ユビキチン修飾にはモノユビキチン化と8種類の異なる構造のポリユビキチン鎖が存在し、それらが使い分けられることで標的タンパク質の運命が決定されます。さらに、最近では混合鎖や分岐鎖など複雑なユビキチン鎖も細胞内に存在することが示唆されています。このようにユビキチン修飾は"ユビキチンコード"とも呼ぶべき膨大な情報を内包しており多彩な生理機能を発揮しますが、ユビキチン修飾の構造すなわちユビキチン鎖の種類や長さ、複雑さを決定する解析法が完全には確立されておらず、ユビキチンシグナル発動の機構は未解明な点が多いのが現状です。そこで、本研究では、ユビキチン修飾の情報を生化学的に解読する方法を確立します。まず、ポリユビキチン鎖のユビキチン間結合について質量分析計を用いて絶対定量する系を確立します。また、ポリユビキチン鎖の長さ及び複雑さの解析技術、さらにアーティファクトを極力排除したユビキチン化試料調製法を開発することで、ユビキチンコードの全貌解明に挑戦します。
関連する代表的な論文:
- Yoshida, Y., Saeki, Y., Murakami, A., Kawawaki, J., Tsuchiya, H., Yoshihara, H., Shindo, M., and Tanaka, K. (2015) A comprehensive method for detecting ubiquitinated substrates using TR-TUBE. Proc. Natl. Acad. Sci. USA. 112, 4630-4635.
- Fukushima, T., Yoshihara, H., Furuta, H., Hakuno F., Luan, J., Duan, C., Saeki, Y., Tanaka, K., Iemura, S.-I., Natsume, T., Chida, K., Nakatsu, Y., Kamata, H., Asano, T., and Takahashi, S-I. (2015) Nedd4-induced mono-ubiquitination of IRS-2 enhances IGF signalling and mitogenic activity. Nature Commun. 6, 6780.
- Morimoto, D., Walinda, E., Fukada, H., Sou, Y.-S., Kageyama, S., Hoshino, M., Fujii, T., Tsuchiya, H., Saeki, Y., Arita, K., Ariyoshi, M., Tochio,H., Iwai, K., Namba, K., Komatsu, M., Tanaka, K., and Shirakawa, M. (2015) The Unexpected Role of Polyubiquitin Chains in the Formation of Fibrillar Aggregates. Nature Commun. 6, 6116.
- Ohtake, F., Saeki, Y., Sakamoto, K., Ohtake, K., Nishikawa, H., Tsuchiya, H., Ohta, T., Tanaka, K., and Kanno, J. (2015) Ubiquitin acetylation modulates polyubiquitin chain elongation. EMBO Rep. 16, 192-201.
- Reincke, M., Sbiera, S., Hayakawa, A., Theodoropoulou, M., Osswald, A., Beuschlein, F., Meitinger, T., Mizuno-Yamasaki, E., Kawaguchi, K., Saeki, Y., Tanaka, K., Wieland, T., Graf, E., Saeger, W., Ronchi, CL., Allolio, B., Buchfelder, M., Strom, TM., Fassnacht, M., and Komada, M. (2015) Mutations in deubiquitinase USP8 cause Cushing's disease. Nature Genetics, 47, 31-38.
- Koyano, F., Okatsu, K., Kosako, H., Tamura, Y., Go, E., Kimura, M., Kimura, Y., Tsuchiya, H, Yoshihara, H., Hirokawa, T., Endo, T., Fon, E.A., Trempe, J., Saeki, Y., Tanaka, K., and Matsuda, N. (2014) Ubiquitin is phosphorylated by PINK1 to activate parkin. Nature 510, 162-166.
- Pack, C.G., Yukii, H., Toh-e, A., Kudo, T., Tsuchiya, H., Kaiho, A., Sakata, E., Murata, S., Sako, Y., Baumeister, W., Tanaka, K., and Saeki, Y. (2014) Quantitative live-cell imaging reveals spatio-temporal dynamics and cytoplasmic assembly of the 26S proteasome. Nature Commun. 5, 3396.
- Takiuchi, T., Nakagawa, T., Tamiya, H., Fujita, H., Sasaki, Y., Saeki, Y., Takeda, H., Sawasaki, T., Buchberger, A., Kimura, T., and Iwai, K. (2014) Suppression of LUBAC-mediated linear ubiquitination by a specific interaction between LUBAC and the deubiquitinases CYLD and OTULIN. Genes Cells 19, 254-272.
- Tsuchiya, H., Tanaka, K., and Saeki, Y. (2013) The parallel reaction monitoring method contributes to a highly sensitive polyubiquitin chain quantification. Biochem. Biophys. Res. Commun. 436, 223-229.
- Saeki, Y., Toh-e, A., Kudo, T., Kawamura, H., and Tanaka, K. (2009) Multiple proteasome-interacting proteins assist the assembly of the yeast 19S regulatory particle. Cell 137, 900-913.