ユビキチンリガーゼによる
選択的基質識別メカニズム


研究概要:
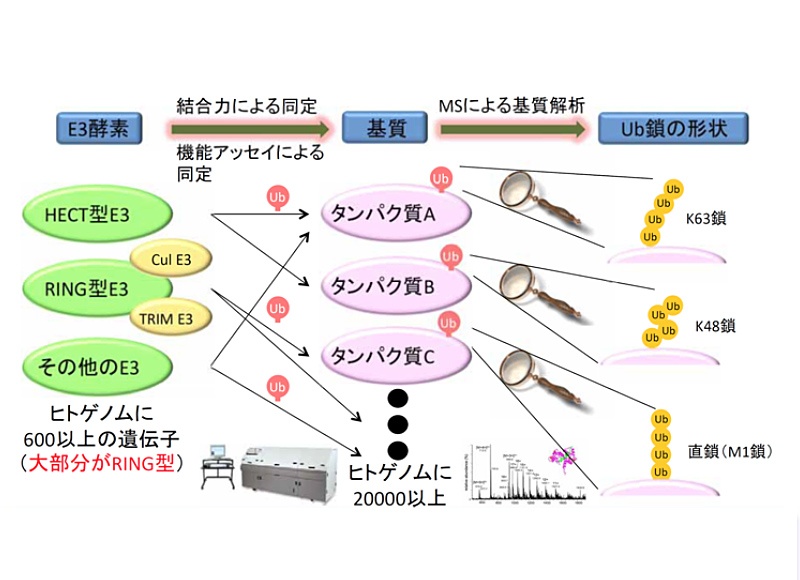
ユビキチン修飾系は選択的にタンパク質を識別し、多彩な形態のユビキチン(ポリユビキチン鎖など)を結合させて分解のみならず多様な様式でその機能を制御することで、細胞周期進行・シグナル伝達・DNA複製・神経変性疾患・免疫応答など多岐にわたる生命現象において重要な役割を果たしています。ユビキチン系はユビキチン活性化酵素(E1)、ユビキチン結合酵素(E2)、ユビキチンリガーゼ(E3)の3種類の触媒酵素群によって、E3が特異的に認識する基質タンパク質に種々の様式のユビキチン鎖を結合させることで始動します。このことより、E3による基質識別メカニズムの理解はユビキチンネオバイオロジーの解明に不可欠な研究テーマです。ユビキチン修飾される標的タンパク質は非常に多く、それを特異的に認識するE3も非常に多数(ヒトで約600種類)存在しています。そこで本研究では、他の計画研究と共同し、様々なE3に対する基質を免疫沈降法および質量分析法の組み合わせにより分離・同定し、さらにはこれら酵素・基質関係により制御される生命現象を解明することを目的とします。
関連する代表的な論文:
(嘉村)
- Nakatsukasa, K., Nishimura, T., Byrne, D., Okamoto, M., Takahashi-Nakaguchi, A., Chibana, H., Okumura, F., and Kamura, T. (2015) The ubiquitin ligase SCFUcc1 acts as a metabolic switch for the glyoxylate cycle. Mol. Cell, doi:10.1016/j.molcel.2015.04.013.
- Nakatsukasa, K., Kanada, A., Matsuzaki, M., Byrne, S.D., Okumura, F., and Kamura, T. (2014) The nutrient stress-induced small GTPase Rab5 contributes to the activation of vesicle trafficking and vacuolar activity. J. Biol. Chem. 289, 20970-20978.
- Sato, Y., Yoshizato, T., Shiraishi, Y., Maekawa, S., Okuno, Y., Kamura, T., Shimamura, T., Sato-Otsubo, A., Nagae, G., Suzuki, H., Nagata, Y., Yoshida, K., Kon, A., Suzuki, Y., Chiba, K., Tanaka, H., Niida, A., Fujimoto, A., Tsunoda, T., Morikawa, T., Maeda, D., Kume, H., Sugano, S., Fukayama, M., Aburatani, H., Sanada, M., Miyano, S., Homma, Y., and Ogawa, S. (2013) Integrated molecular analysis of clear-cell renal cell carcinoma. Nat. Genet. 45, 860-867.
- Nakatsukasa, K., Brodsky, J.L., and Kamura, T. (2013) A stalled retrotranslocation complex reveals physical linkage between substrate recognition and proteasomal degradation during ER associated degradation. Mol. Biol. Cell 24, 1765-1775
- Okumura, F., Okumura, A.J., Uematsu, K., Hatakeyama, S., Zhang, D.E., and Kamura, T. (2013) Activation of double-stranded RNA-activated protein kinase (PKR) by interferon-stimulated gene 15 (ISG15) modification down-regulates protein translation. J. Biol. Chem. 288, 2839-2847
(畠山)
- Tsukiyama, T., Fukui, A., Terai, S., Fujioka, Y., Shinada, K., Takahashi, H., Yamaguchi, T.P., Ohba, Y., and Hatakeyama, S. (2015) Molecular role of RNF43 in canonical and noncanonical Wnt signaling. Mol. Cell. Biol. 35, 2007-2023.
- Sato, T., Takahashi, H., Hatakeyama, S., Iguchi, A., and Ariga, T. (2015) The TRIM-FLMN protein TRIM45 directly interacts with RACK1 and negatively regulates PKC-mediated signaling pathway. Oncogene 34, 1280-1291.
- Takahashi, H., Takigawa, I., Watanabe, M., Anwar, D., Shibata, M., Tomomori-Sato, C., Sato, S., Ranjan, A., Seidel, C.W., Tsukiyama, T., Mizushima, W., Hayashi, M., Ohkawa, Y., Conaway, J.W., Conaway, R.C., and Hatakeyama, S. (2015) MED26 regulates the transcription of snRNA genes through the recruitment of little elongation complex, Nat. Commun. 5, 5941.
- Yaguchi, H., Okumura, F., Takahashi, H., Kano, T., Kameda, H., Uchigashima, M., Tanaka, S., Watanabe, M., Sasaki, H, and Hatakeyama, S. (2012) TRIM67 protein negatively regulates Ras activity through degradation of 80K-H and induces neuritogenesis. J. Biol. Chem. 287, 12050-12059.
- Sato, T., Okumura, F., Ariga, T., and Hatakeyama, S. (2012) TRIM6 interacts with Myc and maintains the pluripotency of mouse embryonal stem cells. J. Cell Sci. 125, 1455-1555.